Het is zo’n bericht dat je “overal” tegen kon komen, maar waar ik toch ook zelf een bericht over wilde schrijven: Microsoft is er in geslaagd om 200MB aan data in synthetische DNA op te slaan en weer uit te lezen.
Het is zo’n bericht dat je “overal” tegen kon komen, maar waar ik toch ook zelf een bericht over wilde schrijven: Microsoft is er in geslaagd om 200MB aan data in synthetische DNA op te slaan en weer uit te lezen.
De omvang van de hoeveelheid DNA die daarvoor nodig was, was minder dan het puntje van een potlood.
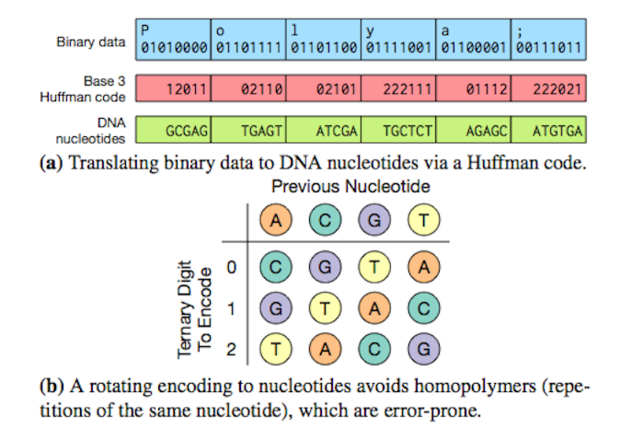
Het bericht op ComputerWorld heeft meer details. Het plaatje hiernaast is afkomstig uit het bericht van eerder dit jaar en laat zien hoe je van een letters/characters, via Binaire data (1-en en 0-en) met een tussenstap via Base 3 Huffman codering bij een codering in DNA nucleotides.
Die Base 3 Huffman codering kende ik nog niet, het is een verliesloze compressie die gebruik maakt van de frequentie van verschijnen van de binaire data. Simpel gezegd: als je alle letters in een brief een unieke code moet geven, dan is het handiger om de letters die het vaakst voorkomen de kortste mogelijke code te geven en de langere codes te gebruiken voor de letters die minder vaak verschijnen in de tekst.
Het tweede deel van het plaatje zorgde ook even voor wat denkwerk: hoe kom je van 12011 => GCGAG. Dat vergde even was puzzelen op de tabel die er onder staat. Nou is het voor de eerste vertaling van 1 => G niet echt eenduidig, want ik weet niet wat de vorige nucleotide was, maar vanaf daar gaat het gemakkelijker. Want het tweede cijfer is 2, als je dan in de rij met ervoor het cijfer 2 kijkt en dan de kolom opzoekt die hoort bij de vorige nucleotide (G in dit geval), dan zie je dat die 2 in dit geval als C wordt gecodeerd. De 0 die volgt wordt dan vastgelegd als G (zie de rij bij de 0 en de kolom C). De daaropvolgende 1 wordt dan een A etc.
Ik kan me dus voorstellen dat zowel het schrijven als lezen van de data niet erg snel gaat. Er komt het nodige rekenwerk bij kijken vooraf bij het encoderen via Base 3 Huffman en de afhankelijkheid van de nucleotides maakt dat fouten best ernstig kunnen zijn. Immers, ook bij het teruglezen is de “betekenis” (0,1,2) van een nucleotide (A,C,G,T) afhankelijk van de vorige nucleotide.
Het blijft echter interessante technologie, en ook harde schijven waren ooit onpraktisch van omvang. Dit is heel klein maar voor dagelijks gebruik ook nog onpraktisch. Dat kan (zal) nog veranderen.